生物信息学 第三章
- 格式:doc
- 大小:89.50 KB
- 文档页数:12



生物信息学知识点总结分章第一章:生物信息学概述生物信息学是一门综合性学科,结合计算机科学、数学、统计学和生物学的知识,主要研究生物系统的结构、功能和演化等方面的问题。
生物信息学的发展可以追溯到20世纪70年代,随着基因组学、蛋白质组学和生物技术的发展,生物信息学逐渐成为生物学研究的重要工具。
生物信息学的主要研究内容包括基因组学、蛋白质组学、代谢组学、系统生物学等。
生物信息学方法主要包括序列分析、结构分析、功能预测和系统分析等。
第二章:生物数据库生物数据库是生物信息学研究的重要基础,主要用于存储、管理和共享生物学数据。
生物数据库包括基因组数据库、蛋白质数据库、代谢数据库、生物通路数据库等。
常用的生物数据库有GenBank、EMBL、DDBJ等基因组数据库,Swiss-Prot、TrEMBL、PDB等蛋白质数据库,KEGG、MetaCyc等代谢数据库,Reactome、KeggPathway等生物通路数据库等。
生物数据库的建设和维护需要大量的人力和物力,目前国际上已建立了众多生物数据库,为生物信息学研究提供了丰富的数据资源。
第三章:序列分析序列分析是生物信息学研究的重要内容,主要应用于DNA、RNA、蛋白质序列的比对、搜索和分析。
常用的序列分析工具包括BLAST、FASTA、ClustalW等,这些工具可以帮助研究人员快速比对和分析生物序列数据,从而挖掘出序列的相似性、保守性和功能等信息。
序列分析在基因组学、蛋白质组学和系统生物学等领域发挥着重要作用,是生物信息学研究的基础工具之一。
第四章:结构分析结构分析是生物信息学研究的另一个重要内容,主要应用于蛋白质、核酸等生物分子的三维结构预测、模拟和分析。
常用的结构分析工具包括Swiss-Model、Modeller、Phyre2等,这些工具可以帮助研究人员预测蛋白质或核酸的三维结构,分析结构的稳定性、功能和相互作用等特性。
结构分析在蛋白质结构与功能研究、蛋白质药物设计等方面发挥着重要作用,为生物信息学研究提供了重要的技术支持。



生物信息学与生物医学工程中的数据挖掘与信息整合方法第一章:引言生物信息学与生物医学工程是生命科学和信息技术的交叉学科领域,其目标是通过利用大规模的生物数据,发现生物学上的模式和关联,并应用于生命科学的研究和医学的实践。
在这个领域中,数据挖掘和信息整合方法起着至关重要的作用。
本文将重点介绍生物信息学与生物医学工程中的数据挖掘与信息整合的方法和应用。
第二章:数据挖掘方法数据挖掘在生物信息学和生物医学工程中被广泛应用。
其中,机器学习是一种常用的数据挖掘方法。
通过对已知的生物数据进行特征提取和分类、回归、聚类等算法分析,可以预测和发现新的生物学模式和关联。
此外,深度学习方法也被应用于处理高维、大规模的生物数据,如基因组数据和蛋白质结构数据。
此外,关联规则挖掘和序列挖掘等方法也被用于生物学序列数据的分析和发现。
第三章:信息整合方法生物信息学与生物医学工程中的数据来自于各种不同的数据源,如基因组学、转录组学、蛋白质组学、代谢组学等。
这些数据源之间的整合是十分重要的。
信息整合方法包括数据标准化、数据集成和数据挖掘等技术。
例如,基因表达数据集成可以通过将不同实验室和平台上的数据整合为一个一致的数据集,从而提高数据的可靠性和一致性。
此外,还可以应用本体论等知识表示方法来实现不同数据源之间的语义一致性。
第四章:应用案例一:生物标志物发现生物标志物是指与某种疾病或生物过程相关的特定分子或生物特征。
生物信息学和生物医学工程中的数据挖掘方法可以用于发现生物标志物。
通过分析大量的生物数据,如基因表达数据、蛋白质组学数据等,可以发现与疾病相关的分子特征。
这些标志物的发现有助于疾病的早期诊断、预测疾病进展和疾病治疗的响应。
第五章:应用案例二:药物研发生物信息学和生物医学工程在药物研发过程中也发挥着重要作用。
数据挖掘方法可以帮助筛选药物靶点、预测药物与靶点的互作、优化药物分子结构等。
通过分析已知的药物分子和靶点的关联数据,可以发现新的药物靶点和药物分子,为药物研发提供新的方向。





生物信息学(上海海洋大学)智慧树知到课后章节答案2023年下上海海洋大学上海海洋大学第一章测试1.生物信息学涉及到以下哪些学科?答案:生物统计学; 生物学;计算机科学2.生物大分子序列里包含了哪些信息?答案:序列信息;功能信息;进化信息;结构信息3.中心法则论述的是遗传信息的流动法则,是指生物大分子的序列决定结构,结构决定功能。
答案:错4.数据是经过加工的信息,对我们做判断和决策有用。
答案:错5.以下哪些观点不是达尔文的《物种起源》提出来的?答案:上帝创造万物6.人类基因组工作草图是什么时候发表的?答案:20017.学好生物信息学最重要的途径是多练习多实践。
答案:对8.世界上最主要的测序公司之一华大基因,是在哪个国家成立的?答案:中国9.以下哪位科学家提出了分子钟假说?答案:泡林 Pauling10.以下哪些组学研究属于生物信息学研究内容?答案:转录组学;基因组学;表观组学;蛋白质组学第二章测试1.以下哪个数据库不是NCBI的子数据库?答案:genecard2.以下哪些数据库属于一级结构数据库?Genbank ;PDB3.在线生物大分子数据库,不可以通过以下哪种方式进行数据查询?答案:电话查询4.在对基因进行查询的时候,如果我们查询的是“cell division[GO]”,我们是通过一下哪种信息对基因进行查询?答案:基因的功能5.蛋白质的profile描述的是具有多个motif的蛋白质家族中,它们具有哪些Motif,以及这些motif的空间分布答案:错6.蛋白质三级结构的实验测定方法包括( )电子显微镜;核磁共振;X光衍射7.ENSEMBL中的gene tree,收集的是同源基因序列答案:对8.KEGG包括以下几类子数据库()答案:chemical information;system information;genomic information; health infromation9.PDB是一个基于功能域进行分类的蛋白质序列数据库。



《生物信息学》第三章:序列比较(第一部分)序列两两比较之打点法:Dotlet界面介绍打点法确实简单,用笔和纸就可以搞定。
到此为止,我知道你一定在怀疑它的实际可操作性。
如果是条长度20的序列,可能你还会硬着头皮拿笔画一画。
如果是条长度200的序列,你一定觉得,老师在逗你玩!没错,没有人真的拿笔画,老师我都是用软件自动完成的。
可以自动打点的软件有很多(表1)。
我们挑其中最常用的Dotlet软件做为演示Dotlet基于Java开发,所以页面打开后会蹦出JAVA对话框。
像对待Jsmol一样,接受JAVA,信任JAVA,运行JAVA。
当然前提是你的电脑已经安装了JAVA。
如果还没有安装,可以到课程附件或者JAVA官网下载安装。
别忘了安装后,重启浏览器,JAVA才能生效。
同样的,IE如果不好使,可以尝试其他浏览器。
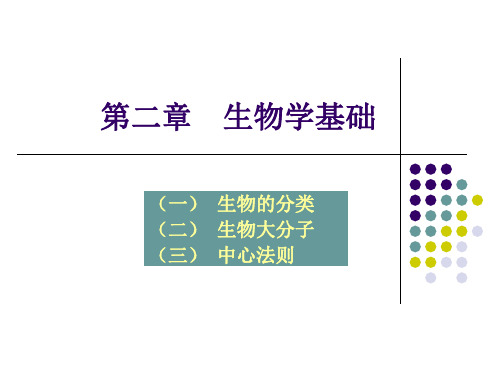
表1. 打点法在线软件网页正常打开后如图1所示。
我们看到上边一行是参数设置选项,包括选择水平位置的序列,选择垂直位置的序列,选择替换记分矩阵,选择以多长的序列为单元打一个点,还有窗口显示的比例,以及计算按钮。
左边是打点图的显示窗口,右边是调控打点图显示效果的区域,下面还有一块是序列信息显示区。
网页打开时除了input按钮,其他位置都是灰的。
图1. Dotlet在线打点工具界面点击input按钮输入要打点的序列。
在新打开的窗口里把要打点的序列拷贝进来。
这里用附件文件dotlet1.fasta里面的序列。
拷贝,黏贴。
注意,序列输入窗口里只能输入纯序列部分。
fasta格式里带大于号的第一行不能拷贝进来,否则Dotlet会把这部分内容也当成序列进行打点。
由于Dotlet不能自动识别FASTA格式里哪是名字,哪是序列,所以我们还需要手动的给这条序列取个名字,比如叫seq1,然后点ok,一条序列就输入进来了。
图1. 打点序列的输入窗口序列输入之后,参数设置行里水平序列的名字和垂直序列的名字都变成了seq1(图2)。
《生物信息学》第三章:序列比较(第二部分)在线双序列比对工具:EMBL局部双序列比对工具EMBL的局部双序列比对工具可以选择经典的Smith-Waterman算法。
仍然比对蛋白质序列。
输入的序列在示例文件local.fasta里面。
More options里面的参数设置和全局比对是一样的。
在这个例子里,我们保持所有参数都为默认值,点提交。
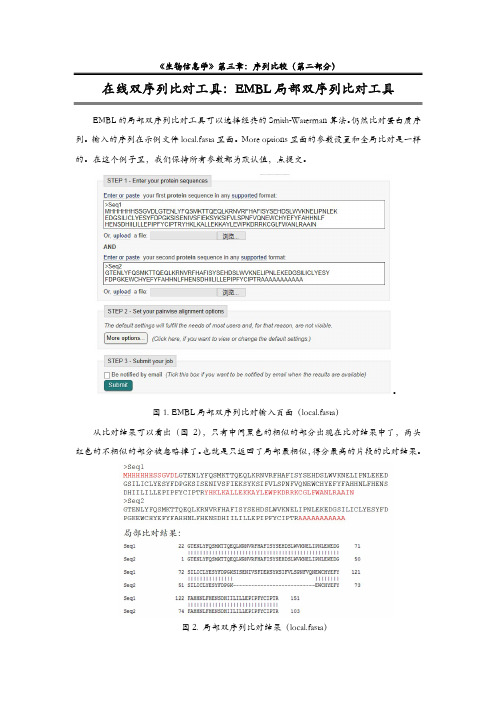
图1. EMBL局部双序列比对输入页面(local.fasta)从比对结果可以看出(图2),只有中间黑色的相似的部分出现在比对结果中了,两头红色的不相似的部分被忽略掉了。
也就是只返回了局部最相似,得分最高的片段的比对结果。
图2. 局部双序列比对结果(local.fasta)用这两条序列再做一次全局比对(图3)。
从两次的比对结果可以更清楚的看出,全局比对里前面和后面对得不好的部分在局部比对里就都被忽略了。
图3. 全局比对与局部比对的比较(local.fasta)除了一长一短两条序列适合做局部比对,有的时候两条差不多长的序列也可以做局部比对,以找出它们最相似的局部片段。
比如示例文件local2.fasta里面的两条序列长度差不多。
我们做局部比对看看结果如何(图4)。
为了让相似的部分突出出来,我们把gap都调大,gap 开头调到10,gap延长调到5,提交。
图4. 局部双序列比对输入页面(local2.fasta)比对结果中,只有黑色的相似的部分出现在最终的比对结果中了,两头红色的不相似的部分全部被忽略了(图5)。
图5. 局部双序列比对结果(local2.fasta)如果给这两条序列做全局比对的话,会发现,绝大部分位置对得都很差,只有中间这一段对的还不错(图6)。
所以,有时候两条序列并不同源,它们只是有一个功能相似的区域,这时用局部比对我们就能很快找到这一区域在两条序列中的位置。
但是如果做全局比对的话,结果就不如局部比对明显了。
图6. 全局比对与局部比对的比较(local2.fasta)。

第一章测试
1
【多选题】(2分)
随着人类基因组计划的完成,以下哪些基因组计划是近期启动的计划
A.
英国十万人基因组计划
B.
G10K
C.
我们所有人计划
D.
中国十万人基因组计划
2
【判断题】(2分)
统计学是一门独特学科,不是生物信息学研究工具和手段之一。
A.
错
B.
对
3
【判断题】(2分)
生物信息学研究任务之一包括SNP的发现和鉴定,对于疾病机理和药物开发靶点发现具有重要意义。
A.
对
B.
错
4
【判断题】(2分)
随着越来越多大规模测序项目的完成,其中最重要的科学使命之一就是要通过比较基因组学方法了解物种的起源和进化过程
A.
错
B.
对
5
【判断题】(2分)
高等生物基因组中含有大量的非编码区,以及可能含有大量的外源病毒序列,只有通过生物信息学方法,解析其中功能和区域,为将来可能通过基因组编辑技术进行疾病机制解析提供基础
A.
对
B.
错
第二章测试
1
【多选题】(2分)
国际核酸数据库由EMBL,DDBJ和GenBank组成,它们在1988年形成国际核酸数据库联合中心,对数据进行
A.
三方共享
B.
数据同步更新
C.
数据格式相同
D.
独立分析
2
【多选题】(2分)
GenBank对于核酸数据的显示方式有以下几种
A.
Graph
B.
ASN.1
C.
GBK
D.
FASTA。
第三章结构数据库【前介】本章将集中介绍生物信息学中生物分子结构的有关内容,并将研究重点放在三维结构实际存在的氨基酸序列上,力图使读者了解结构数据库记录的内容及如何合理应用各类通用软件程序处理这类记录。
本章不涉及结构生物学家们建立三维分子结构的计算程序,也不讨论相似蛋白质构象的精细结构。
在本章参考书目后列出了一些优秀的讨论蛋白质构象的有关专著和蛋白质结构决定方法。
用图象直观表示蛋白质和核酸结构在生物化学教科书和研究论文中屡屡出现。
这些图象是美丽迷人的反而使我们忽视了图象背后所反映的实验细节���实验中应用的生物物理方法,X射线晶体衍射学家和核磁共振波谱分析学家们努力工作的成效.在结构数据库中记录的数据是实用化的实验数据。
它既不同于直接由仪器获得的原始数据,也并非原始数据的简单数学转换。
每一个结构数据库记录都内含着随结构预测技术的进步而不断变化的假设和偏好。
尽管如此,每个生物分子结构蕴涵着有关序列所缺失数据的至关重要的信息。
∙三维分子结构数据的一些概念首先做一个关于如何记录生物高聚物的三维数据的思想实验。
考虑一下如何在纸上记录如肌球素这类蛋白质的三维球棒模型的所有细节和尺度关系。
一条开始的途径是从由三维模型主干描绘出的氨基酸序列入手。
从N’端开始,我们通过将每个残基的化学结构与20种普通氨基酸化学结构(其结构的图解可以从教科书中找到)比较,以识别每个氨基酸侧链。
一旦序列被写出来,我们将绘制生物高聚物的二维草图,草图中包括所有的原子、基本符号、化学键,可能会占用几页纸。
亚化血红素配合基的绘制即为一例。
将它的化学结构画在纸上后,我们可以通过量测模型中每个原子在设定的直角坐标系中的距离记录三维数据。
同时也提供了球�棒结构中每个原子“球”的x,y,z坐标距离数据。
下一步是提出一个系统的分门别类的记录方案以保存与识别有关的每个原子的(x,y,z)坐标信息。
最简单的方法是在生物高聚物的二维草图上,每个原子的右侧,标出(x,y,z)三元坐标值。
以上思想实验有助于我们对三维结构数据库应包含哪些内容形成初步的概念。
从人类可读性的角度而言,这样的结构记录形式是足够的,但计算机却不一定能够理解它。
计算机需要原子、化学键、坐标、残基、分子间结合关系的清晰明显的编码。
∙坐标、序列、化学图像典型的三维结构记录中与使用的文件格式无关的最明显的数据是坐标数据,它表征了分子中原子的空间位置,用沿着每个坐标轴到某特定原点的距离(x,y,z)表示。
每个原子的坐标数据归属于结构记录中的标注信息列表:空间中的每一点代表了记录中的元素、残基和分子。
对于生物多聚体,这标注信息来源于序列。
每个序列固含的是重要的化学数据。
我们能够直接从序列中推断出完整的包含所有原子和化学键的生物高聚物分子化学联接,能够恰如早先所描述的仅从序列信息出发描绘出草图。
我们把这分子“草图”称作三维结构的化学图像。
序列是生物高聚物分子完整化学图谱的固有表示。
当描绘从属原子与化学键的略图以表示序列时,我们可以借鉴教科书中描绘的每个残基的化学结构,以免露掉一两个甲基。
同样地,计算机可利用“残基词典”在内存中建立结构的类似于略图的化学图像表示,“残基词典”中则包括一组对应于每个普通氨基酸或核酸模块的原子类型与化学键信息表。
原子、化学键和完整性分子图像可视化软件完成了精细的“点联接”过程,而绘制出如我们在生物分子结构教科书中所见到的完美的蛋白质结构图像,例如图3.1所示的胰岛素3INS 结构(Isaacs,Agarwal,1978)。
显然,原子间联接依靠化学键。
在目前的应用中,三维分子结构数据库记录使用了两种不同的键数据信息优化存储方法。
记录原子与化学键信息的经典途径是依靠“化学准则”。
这些准则是显而易见的物理化学准则,比如稳定的碳、碳键的平均长度大约1.5埃。
应用这些来源于化学键的规则,意味着空间中两个1.5埃距离的碳原子总形成单键。
有了这些化学准则,我们可完全简化化学键信息存储。
倘若结构本身未违背任何化学规则,则能够被完整记录而不带任何附加键信息。
最初的三维生物分子结构文件记录格式,Brookhaven蛋白质数据库(Bernstein 等,1977)的PDB格式皆以化学准则方法为基础。
一般而言,这些记录没有生物高聚物的完整键信息。
无需“残基词典”,而仅用可能成键原子对的键长与键类型匹配表即可解译用“化学准则方法”编码的数据。
PDB数据文件读入软件包必须能基于这类规则重构化学键。
对于程序员,如何解释PDB文件中的键信息尚未形成明确统一的规则,而导致了各类软件绘制化学键连接时的不一致,尤其应用了不同的算法和距离容差,这类情况更为严重。
虽然PDB文件组织方案在记录数据存储方面的要求最低,但比较连键信息和化学图像描述已在记录中详细说明的情况,则对存储信息进行恰当解释所需算法也相应更复杂。
这将迫使程序开发者做更多的工作。
基于事件的编程中,考虑连键规则中的种种例外情况,更需要复杂的逻辑说明。
第二种方法在由PDB衍生而来的分子建模数据库(MMDB)的数据库记录中得到应用。
MMDB运用标准的“残基词典”,其中记录了氨基酸、核酸残基这样以聚合体形式存在,具有末端多样性的分子中所有原子、化学键信息。
在结构科学家解决分子结构而使用的专用软件中,这类数据词典是很普遍的。
读入MMDB数据的软件能利用词典所提供的键信息将原子连为一体,而无须力图满足化学准则的要求。
最终,用软件获得准确的三维坐标数据。
这种方法使软件开发简单化,因为连键规则中的例外情况在数据库文件中已被记录,而无须附加逻辑控制代码即可将之读入。
一些不熟悉结构数据的科学家常常希望在公共数据库中的结构信息表达类同于教科书。
他们会对结构中某部分的数据丢失感到惊讶。
相应于某一特定分子的三维数据库记录的适用性并不意味着完整性。
结构的完整性定义如下:化学图像中任一原子至少有一维坐标值确定。
在结构数据库中,完整的记录是不多见的。
大多数由X射线衍射获得的结构缺少氢原子坐标,因为氢原子的空间位置不能用实验手段决定。
但一些建模软件可用于估计氢原子位置,并用其重建结构记录。
在结构数据库中识别由模型构造的分子是容易的。
它们常常有过于复杂的坐标数据和所有用实验手段无法确认的氢原子可能表达形式。
【PDB:Brookhaven国家实验室蛋白质数据库】∙概述计算机在生物学中的运用起源于生物物理方法的应用,如X射线结晶衍射。
于是最初的“生物信息学”数据库被用于存储复杂的三维数据不足为怪。
现代的蛋白质数据库以收集的蛋白质三维结构公共数据为核心,附带核酸、糖类三维结构和各类由X射线衍射结晶学家、核磁共振谱分析学家通过实验测定的合成物。
本部分集中详细介绍由蛋白质数据库PDB提供的生物信息学数据库服务。
∙PDB数据库服务Brookhaven国家实验室(详见本章末列表)蛋白质数据库的WWW站点为三维结构数据的提交、检索提供了大量的服务。
∙提交结构数据对于那些希望向PDB提交三维结构信息的人们而言,可以经由AutoDep服务机构按照一定的基于网页的程序步骤实现其愿望。
因为提交程序是随编写时间而不断变化的,所以在PDB的网络站点上应该能找到最新信息。
核酸结构数据保存在核酸数据库NDB中。
Biotech Validation Suite站点是镜像站点,提供在提交结构数据前屏蔽立体化学构象与几何学构象不一致的PDB文件的服务。
PDB明文规定拒收依靠计算机三维建模而非实验手段获得的结构数据。
而关于已被宣布为例外结构的最新细节数据的提交需与PDB商议。
容纳结构模型的单独的数据库是现成的,可以在本书的网络站点上查询有关信息。
∙PDB的ID编码PDB中登记入册的结构记录拥有一个唯一的包含字母与数字的被称为PDB-ID或PDB编码的四位字符串,可由数字0~9和大写字母A~Z组合而成。
因此可能的组合方案超过了130万种,没有按某特定顺序分配PDB-ID。
但蛋白质数据库PDB 的索引编撰者尽量设计好的记忆方法,使结构名称易于记忆,如早先如图3.1所示的胰岛素记录3INS。
∙数据库查询、PDB文件检索与链接PDB和它的一些镜像站点提供由每个PDB记录的所有文本信息索引的文本搜索引擎,可按一些专门的查询项目(如提交数据、作者姓名、结构表达)检索。
PDB 最新的搜索引擎,3DB Atlas,可用于PDB记录检索,如图3.2示。
3DB Atlas 也是链接有PDB结构数据第三方注解的基本数据库,支持大量的到基于因特网三维结构服务的其它网点的链接。
其中包括了一些二维、三维浏览器,如Kinemage (Richardson,Richardson,1992)、Resmol(Sayle,Milner�White,1995)。
图3.2b显示了蛋白质1BNR的到3DB记录Barnase的一些链接。
创建的图像有助于调整三维结构方向,以获得观察结合位点这类确定特征的最好视角。
3DB Atlas 也与专门设计的数据库相连,这些数据库由对诸如结构进化(FSSP:Holm,Sandar,1993)、结构相似性(DALI:Holm,Sander,1996)和蛋白质运动(Gerstein 等,1994)等相关课题有兴趣的研究者维护。
3DB可相应链接NCBI的MMDB服务(Hogue等,1996),提供了一条到Entrez(Schuler等,1996)系统(包括序列、分类、PubMed/MEDICINE服务和VAST结构相似性比较)的通路。
∙源自PDB结构记录的序列PDB文件编码格式的序列是众人皆知的。
因为不能确保结构的完整,PDB记录包括两个序列信息备份:隐性序列和显性序列。
两者都被用于重构生物高聚体的化学图像。
显性序列在PDB文件中以关键词SEQRES打头逐行存储。
不同于其它序列数据库,PDB记录用三字母氨基酸编码,任意选择三个字母作为名称的非标准氨基酸在许多PDB记录序列条目中可被找到。
在PDB中,一些双螺旋核酸序列条目被指定依照在条目中按从3’到5’端的顺序排列的一条链在上,从5’到3’端排列的互补链在下的方式排列。
虽然这些以双螺旋形式表达的序列对人类而言是容易理解的,但直接由计算机阅读此类从3’到5’端排列的显性序列是荒堂的。
因为三维结构可能对应有多个生物高聚物链,所以使用者必须借助PDB链识别标记方可确定需要的序列。
PDB文件SEQRES入口用一个大写字母或空格作为链识别标记,以识别条目中的每个单独的生物高聚体链。
如图3.1所示的3INS结构,在记录中便存在两种胰岛素分子。
3INS序列包括A、B、C、D四个氨基酸序列。
由胰岛素的生物化学背景知识知道A、B蛋白质链源自同一基因,在翻译修饰的过程中,胰岛素序列被切为如PDB记录所示的两段。